Nachweis der Kopplung durch Stammbaumanalyse
Beispiel: Gleichzeitige Vererbung von Rot-Grün Farbenblindheit (p)
und Hämophilie (h) mit Crossing-over!

3. Generation: Das 1. Kind trägt 2 Gendefekte, welche sich phänotypisch
auswirken! Da die Defekte rezessiv sind, müssen sie homozygotisch sein.
Die Mutter muss Konduktorin sein, d. h. sie trägt das rezessive Gen
heterozygot (einmal). Da der Sohn vom Vater nur ein Y bekommt, müssen
die defekten Gene beide von dem von der Mutter abgegebenen X-Chromosom stammen!

X-chromosomale Genkarte (durch Kopplungsanalyse erstellt)
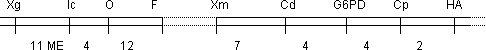
Xg = Blutgruppe, Erythrozyten-Antigen Xg+ (man hat es)
oder Xg- (man hat es nicht)
Ic = Ichthyosis (Fischschuppenkrankheit, X-chromosomal
rezessiv); bei Männern (selten bei Frauen), extrem ledrige Haut
O = Okularer Albinismus (nur das Auge ist pigmentlos),
das Auge erscheint rot, da durch das Fehlen der Pigmente die Kapillaren
sichtbar werden
F = Fabry disease: Mangel an Glucolipid-Lipidasen ( ›
Lipidanlagerung in der Blase)
Xm = Serum-Eigenschaft
Cd = Deutan-Locus (grün-farbenblind)
G6PD = Glucose-6-Phosphat-Dehydrogenase
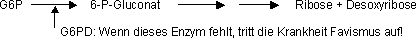
Favismus: Lat. faba, ital. Fava: Bohnen › Patienten
vertragen weder Bohnen noch Aspirin oder Malariamedikamente (Primaquine),
sie zeigen eine schwere hämolytische Reaktion (hämolytische Anämie
mit Hämoglobinurie), eine Bluttransfusion wird notwendig. Die Krankheit
kommt vor allem in der Region Mittelmeer vor (Italien) und vor allem bei
Männern.
Cp = Protan-Locus (rot-farbenblind)
HA = Hämophilie A
Heute sind auf dem X-Chromosom ca. 300 Gene bekannt, ermittelt mit Hilfe
verschiedener Methoden.
Autosomale Kopplungsgruppen:
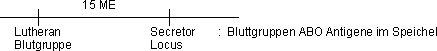
Der Nachweis autosomaler Kopplungsgruppen ist beim Menschen schwierig,
da nur geringe Kinderzahlen pro Ehe existieren und keine Experimente machbar
sind.
Genkartierung durch Deletion
- Blutgruppe MN (diese Unterscheidung ist im Gegensatz zu ABO-System
nicht relevant)
- Vererbung autosomal
- kodominant (d.h. wir haben 3 Genotypen und wegen der Kodominaz auch
3 Phänotypen!!)
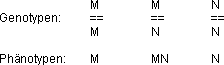
Man untersuchte den Stammbaum eines Knaben, der hemizygot für den
autosomalen Blutgruppenlocus MN war.
Hemizygotes Gen: Das ist ein Gen, das allein und nicht in der Form eines
Allelpaares auftritt! Männer sind bezüglich aller X-chromosomalen
Gene hemizygot.
Ein Mensch wird ebenfalls hemizygot für ein Gen, dessen Allel durch
Chromosomenstückverlust (Deletion) augefallen ist.
Stammbaumanalyse:
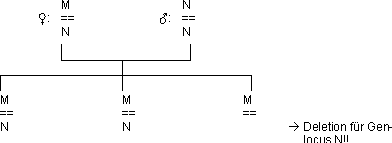
Zu erwarten wäre bei den Nachkommen 50 % M//N und 50 % N//N; der Nachkomme
M// dürfte nicht sein.
Die cytologische Untersuchung der Blut-Lymphocyten ergab, dass eine reziproke
Translokation zwischen dem langen Arm des 2. Chromosoms und dem langen Arm
des 4. Chromosoms stattgefunden hat. Dabei ist durch Deletion das kleine
Stück 2q14 verlorengegangen:
46, XY, t(2q-; 4q+) und Deletion von 2q14 › Nicht
zwei, sonder drei Brüche!
Anmerkung: Dieser Knabe hat sich nicht normal entwickelt. Wahrscheinlich
waren noch wichtige Informationen auf dem durch Deletion verlorengegangenen
Stück. Im weiteren ist auch die Genbalance gestört.
Transokation:
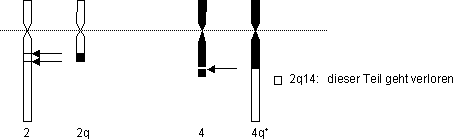
Chromosomenfragment geht während der reziproken Transokation verloren
› Deletion für 2q14
Es handelt sich also um eine reziproke Transokation mit Verlust von Band
2q14.
Schluss: Der Genlocus für MN liegt auf dem Chromosom
Nr. 2 an Position q14!
Ein genetischer Defekt und eine cytologische Deletion wurden verwendet,
um einen Lucus zu bestimmen.
Vorteil: Man kann ein Gen einem bestimmten Chromosom zuordnen und sogar
dessen Lage innerhalb des Chromosoms bestimmen.
Nachteil: Das ganze ist zufallsbedingt!
Translokation: Das Bruchstück eines Chromosoms ist an ein nicht-homologes
Chromosom angeheftet; es sind nachher 2 umgebaute Chromosomen.
Beim Umbau kommt es in der Meiose zu Schwierigkeiten bei der Paarung, da
die Chromosomen nicht mehr auf der ganzen Länge homolog sind. Es kommt
zu einer Kreuzkonjugation, welche es den homologen Regionen erlaubt, nebeneinander
zu liegen zu kommen ( und so C.O.‘s ermöglicht).
Somatische Zell-Hybridisierung
Hybridisierung = Bastardisierung
Versuch:
Man züchtet menschliche Zellen und Mauszellen in vitro, z. B. Fibroblasten,
Lymphocyten, Leukocyten....
Dazu gibt man z. B. abgetötete Sendai-Viren oder Lysolecithin, um die
Zellwand „aufzuweichen“ und so eine Fusion zu erleichtern.
Bei der Fusion von menschlichen und tierischen Zellen entsteht eine Heterokaryonzelle!
HAT-Trick
Voraussetzungen:
- Gen T sorgt für die Weiterverarbeitung von exogenem Thymidin (Pyrimidin-Base)
- Gen H sorgt für die Inkorporation von Hypoxanthin (Purin-Base)
- Mutante T-: Thymidin kann nicht weiterverarbeitet werden, weil Thymidin-Kinase
fehlt.
- Mutante H-: Hypoxanthin kann nicht inkorporiert werden, weil das Enzym
Hypoxanthin-Guanin-Phosphoribosyl-Transferase (HGPRT) fehlt.
HAT = Hypoxanthin, Aminopterin, Thymidin
Aminopterin: blockiert die Eigen- oder Neusynthese von Purinen und Pyrimidinen
HAT-Trick:
theoretisch: nur die fusionierten Zellen können überleben, die
nicht-fusionierten sterben, weil sie mind. einen Defekt haben.
konkret: dies funktioniert nur durch die Wahl eines geeigneten Mediums:
HAT
Menschliche Zelle T- H+
Das exogene (im Kulturmedium enthaltene) Thymidin, welches für die
DNA unentbehrlich ist, kann nicht weiterverarbeitet werden.
Aminopterin verhindert die Neusynthese von Thymidin
Thymidin fehlt für die DNA
Tod
Mauszelle H- T+
Das im Medium enthaltene Hypoxanthin kann nicht inkorporiert werden.
Aminopterin verhindert die Neusynthese von Hypoxanthin
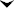
Hypoxanthin fehlt für DNA
Tod

Nach dem programmierten Verlust oder der Segregation von menschlichen Chromosomen
überlebt die Hybridzelle nur, solange das Gen H+ vorhanden ist.
› Es entstehen verschiedene Klone mit vollständigem
Mausgenom, jedoch unterschiedlicher Zusammensetzung menschlicher Chromosomen.
› Isolation von Teilen des menschlichen Genoms
(Möglichkeit zur Genlokalisation)
In unserem Beispiel: Die Hybridzelle überlebt nur, wenn das menschliche
X-Chromosom vorhanden ist.
› Schluss: Das Gen H+ liegt auf dem X-Chromosom
Chromosomenkarten der Menschen
Man nimmt an, dass der Mensch ca. 80'000 Gene besitzt. Bis 1989 waren 5'000
Gene bekannt, 1‘500 konnten auf den Chromosomen lokalisiert werden.
Die Forschung wird seither rasant vorangetrieben und man entdeckt pro Woche
ca. 2 neue Genloci!
Das menschliche Genom:
Lokalisation:
- vorwiegend im Zellkern (Durchmesser ca. 5 µm)
- in 2 x 23 Chromosomen
Genomgrösse:
- ca. 3 x 109 Basenpaare pro haploides Genom, das entspricht etwa 1 m
DNA
- 80'000 Gene, davon 5'000 lokalisiert
- im Körper befinden sich 200 x 109 km Genmaterial (1 m pro Zelle),
das entspricht drei mal der Distanz Erde - Sonne; der Mensch hat
in etwa 6 x 1012 Zellen
Bedeutung der Genlokalisierung:
- Grunlagenforschung
Erlangen von allgemeinem Wisse
- Evolution
Genkarten bei verwandten Arten sehen sich sehr ähnlich, z. B. findet
sich das Gen Enolase I auf dem Chromosom Nr. 1 beim Affen und beim Menschen.
Oder ganze Gencluser (Gengruppen, Gene in der gleichen Reihenfolge) kommen
bei Mensch, Affe und Maus gemeinsam vor.
- Verstehen von Genbalancestörungen
Beispiel: Trisomie 21; vermutlich werden die Störungen hervorgerufen
von einer zu hohen Konzentration des Enzyms Superoxid-Dismutase (SOD).
Diese Überkonzentration verursacht den abnormen Phänotyp durch
Störung des Metabolismus. Im Gegenteil zu beispielsweise Punktmutationen
fehlt kein Genmaterial, es ist im Gegenteil sogar zu viel da.
- Genetische Beratung, pränatale Diagnose
z. B. Diagnose auf Hämophilie A: Das HA-Gen ist im Foetus inaktiv,
es ist also vorderhand nicht möglich festzustellen, ob das Kind hämophil
wird oder nicht. Doch man weiss, dass HA mit G6PD gekoppelt ist und 6
ME auseinander liegen (die Wahrscheinlichkeit für ein Crossing over
beträgt 6 %).
Betrachtet man nun den Stammbaum der schwangeren Frau:
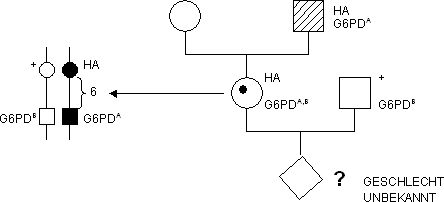
Erklärung: Es gibt zwei Varianten für G6PD: G6PDB = Wildtyp,
G6PDA = Mutante
Der Vater der schwangeren Frau ist hämophil und G6PDA, ihre Mutter
ist gesund (G6PDA fungiert als „Markierung“).
Die Frau ist also heterozygot für Hämophilie und G6PDA, sie ist
also Konduktorin. Ihr Mann ist gesund. Es besteht also die Wahrscheinlichkeit
von 50 %, dass ihr Sohn das X-Chromosom mit HA und G6PDA bekommt, also hämophil
sein wird.
Durch Amniozentese konnte nachgewiesen werden, dass das Kind (Knabe) G6PDB
besitzt. Die Wahrscheinlichkeit, dass der Junge trotzdem hämophil ist,
was nur durch ein Crossing over in der Mutter zwischen HA und G6PDA zustandegekommen
sein kann, beträgt nun also 6 % (den Abstand in ME zwischen den zwei
Loci ist die C. O.-Wahrscheinlichkeit). Die Chance, dass die Frau einen
gesunden Knaben zur Welt bringen wird, beträgt 94 %!
|
